Chapitre 6 Première manipulation des données
library("dplyr")6.1 Afficher les valeurs et manipuler les variables
Pour afficher la table, plusieurs façons : “clic” dans l’environnement Rstudio,
View(base),print(base),base.Pour accéder à une variable : fonction
pull()
Par exemple :
str(pull(base, DEP))## chr [1:36689] "01" "01" "01" "01" "01" "01" "01" "01" "01" "01" "01" "01" ...6.2 Créer de nouvelles variables
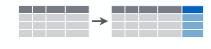
La fonction mutate() permet de créer/modifier une variable (ou plusieurs).
TableEnSortie <- mutate(TableEnEntree,
NouvelleVariable = DefinitionDeLaVariable)Par exemple :
base <- mutate(base, log_SUPERF = log(SUPERF))Nb : mutate() permet également de modifier une variable. Dans ce cas la syntaxe est la même que ci-dessus, mais les noms d’entrée et de sortie sont les mêmes :
base <- mutate(base, log_SUPERF = 100 * log_SUPERF)\(\Rightarrow\) La table base contient de nouvelles colonnes
6.3 Sélectionner des variables
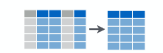
La fonction select() permet de sélectionner les variables voulues.
- sélection par liste blanche
TableEnSortie <- select(TableEnEntree, Variable1, Variable2, ..., VariableN)- sélection par liste noire (supprimer)
TableEnSortie <- select(TableEnEntree, -Variable1, -Variable2, ..., -VariableN)Par exemple :
base_select <- select(base, CODGEO, LIBGEO, P14_POP)
base_select <- select(base, -CODGEO)6.4 Filtrer des observations
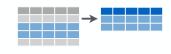
La fonction filter() permet de sélectionner les observations, selon une condition (ou plusieurs).
TableEnSortie <- filter(TableEnEntree, Condition1, ..., ConditionN)Par exemple :
base_filter <- filter(base, DEP == "01" & P14_POP > 10000)\(\Rightarrow\) Attention à l’opérateur de comparaison : ” == ” et non pas “=”
6.5 Les tests logiques dans R
| Syntaxe | Action |
|---|---|
== |
Test d’égalité |
!= |
Différent de |
%in% c(...) |
Dans une liste de valeurs |
>, >= , <, <= |
Supérieur (ou inférieur) (ou égal) |
! (x %in% c(...)) |
N’est pas dans une liste de valeurs |
TableEnSortie <- filter(TableEnEntree, x==a & y==b) # x vaut a **ET** y vaut b
TableEnSortie <- filter(TableEnEntree, x==a | y==b) # x vaut a **OU** y vaut b (barre verticale AltGR+6)6.6 Renommer des colonnes
La fonction rename() permet de renommer une variable (ou plusieurs).
base <- rename(base, nouveau_nom = ancien_nom)Exemple
base_rename <- rename(base, ZONE_EMPLOI = ZE)6.7 Exercice 3 : créer, filtrer, sélectionner
Nous travaillons toujours sur la même table des indicateurs au territoire, “extdata/Base_synth_territoires.csv”.
A- En utilisant la fonction mutate(), créer une nouvelle variable correspondant à la densité de population (rapport de la population P14_POP à la superficie de la commune SUPERF), ainsi que les taux de natalité et de mortalité (en pour mille)
B- A l’aide de la fonction select(), créer une nouvelle table en ne conservant que le code commune, le type de commune (ZAU), la région, le département et les variables que vous venez de créer.
C-Enfin, ne conserver les communes correspondant à votre département de naissance et stocker ce dataframe. Attention au type de la variable département !
D- Avec les opérateurs logiques, faire des essais pour sélectionner des échantillons différents.
Résultat attendu :
6.8 Nom d’un pipe %>% !
Pour enchaîner des opérations, on peut créer des variables successives :
df <- mutate(base, densite = P14_POP / SUPERF,
tx_natal = 1000 * NAISD15 / P14_POP,
tx_mort = 1000 * DECESD15 / P14_POP)
selection <- select(df, CODGEO, ZAU, REG, DEP, densite, tx_natal)
filtre_62 <- filter(selection, DEP == "62")Cela peut être relativement clair si on respecte de bonnes pratiques de nommage mais encombre inutilement l’environnement de travail.
On peut emboîter les fonctions :
selection_62 <- filter(select(mutate(base, densite = P14_POP / SUPERF,
tx_natal = 1000 * NAISD15 / P14_POP,
tx_mort = 1000 * DECESD15 / P14_POP),
CODGEO, ZAU, REG, DEP, densite, tx_natal),
DEP == "62")Ce choix préserve l’environnement de travail mais la lecture est particulièrement confuse et le risque d’oubli de parenthèses important.
Il y a enfin une solution du package magrittr faisant partie du tidyverse.
On peut combiner les opérations en une seule ligne à l’aide de l’opérateur pipe %>% :
selection_62 <- base %>%
mutate(densite = P14_POP / SUPERF,
tx_natal = 1000 * NAISD15 / P14_POP,
tx_mort = 1000 * DECESD15 / P14_POP) %>%
select(CODGEO, ZAU, REG, DEP, densite, tx_natal) %>%
filter(DEP == "62")Cette écriture permet d’enchaîner les opérations telles qu’on les décrirait à l’oral. L’objet auquel s’applique chaque nouvelle opération est le résultat de l’opération précédente.