Chapitre 5 Manipuler des données
5.1 Les principes des fonctions de {dplyr}
Le but de {dplyr} est d’identifier et de rassembler dans un seul package les outils de manipulation de données les plus importantes pour l’analyse des données. Ce package rassemble donc des fonctions correspondant à un ensemble d’opérations élémentaires (ou verbes) qui permettent de :
- Sélectionner un ensemble de variables :
select()
- Sélectionner un ensemble de lignes :
filter()
- Ajouter/modifier/renommer des variables :
mutate()ourename()
- Produire des statistiques agrégées sur les dimensions d’une table :
summarise()
- Trier une table :
arrange()
- Manipuler plusieurs tables :
left_join(),right_join(),full_join(),inner_join()…
D’appliquer cela sur des données, quel que soit leur format : dataframes, data.table, couche spatiale, base de données sql, big data…
D’appliquer cela en articulation avec group_by() qui change la façon d’interpréter chaque fonction : d’une interprétation globale sur l’ensemble d’une table, on passe alors à une approche groupe par groupe : chaque groupe étant défini par un ensemble des modalités des variables définies dans l’instruction group_by().
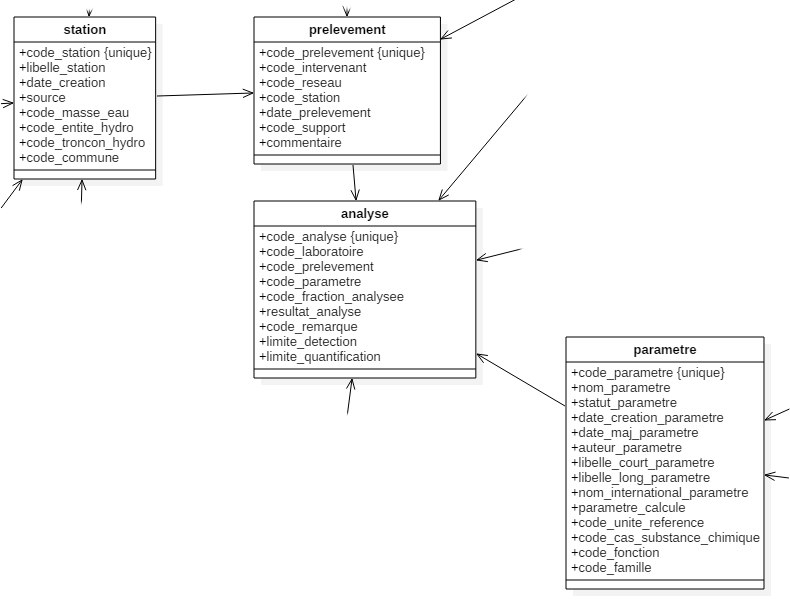
5.2 Présentation des données
On va travailler sur ce module principalement à partir des données sitadel en date réelle estimée (permis de construire) et à partir des données de qualité des eaux de surface.
5.4 Les verbes clefs de {dplyr} pour manipuler une table
5.4.1 Sélectionner des variables : select()
Nous allons ici sélectionner un ensemble de variables de la table des prélèvements.
prelevementb <- select(
prelevement, date_prelevement, code_prelevement,
code_reseau, code_station
)
datatable(head(prelevementb))prelevementb <- select(prelevement, -code_support)
names(prelevementb)## [1] "code_prelevement" "code_intervenant" "code_reseau" "code_station"
## [5] "date_prelevement"select() possède ce qu’on appelle des helpers qui permettent de gagner du temps dans l’écriture de notre sélection.
A partir du moment où les conventions de nommage sont correctement effectuées, cela permet de gagner également en reproductibilité d’une année sur l’autre.
Exemple : sélectionner toutes les variables qui commencent par “code_” :
prelevementb <- select(prelevement, starts_with("code_"))Exemple : sélectionner les variables dont les noms sont contenus dans un vecteur de chaînes de caractères :
mes_variables <- c("code_prelevement", "code_intervenant", "code_reseau", "date_prelevement")
prelevementb <- select(prelevement, one_of(mes_variables))5.4.3 Renommer une variable : rename()
prelevementb <- rename(prelevementb, date_p = date_prelevement)On peut aussi directement renommer une variable dans l’opération select()
prelevementb <- select(prelevement, date_p = date_prelevement, code_prelevement,
code_reseau, code_station)5.4.4 Filtrer une table : filter()
On va ici récupérer les analyses produites par l’ARS
ars <- filter(prelevement, code_reseau == "ARS")L’exemple ci-dessus n’exerce un filtre que sur une condition unique.
Pour des conditions cumulatives (toutes les conditions doivent être remplies), le "&" ou la ","
ars <- filter(prelevement, code_reseau == "ARS", code_intervenant == "44")Pour des conditions non cumulatives (au moins une des conditions doit être remplie), le “|”
ars <- filter(prelevement, code_reseau == "ARS" | code_reseau == "FREDON")Si une condition non cumulative s’applique sur une même variable, privilégier un test de sélection dans une liste avec le %in%
ars <- filter(prelevement, code_reseau %in% c("ARS", "FREDON"))Pour sélectionner des observations qui ne répondent pas à la condition, le ! (la négation d’un test)
Toutes les observations ayant été réalisées par un autre réseau que l’ARS :
non_ars <- filter(prelevement, code_reseau != "ARS")Toutes les observations ayant été réalisées par un autre réseau que l’ARS ou FREDON :
ni_ars_ni_fredon <- filter(prelevement, !(code_reseau %in% c("ARS", "FREDON")))5.4.5 Modifier/ajouter une variable : mutate()
mutate() est le verbe qui permet la transformation d’une variable existante ou la création d’une nouvelle variable dans le jeu de données.
Création de nouvelles variables :
prelevementb <- mutate(prelevementb,
code_prelevement_caract = as.character(code_prelevement),
code_reseau_fact = as.factor(code_reseau)
)Modification de variables existantes :
prelevementb <- mutate(prelevementb,
code_prelevement = as.character(code_prelevement),
code_reseau = as.factor(code_reseau)
)mutate() possède une variante, transmute(), qui fonctionne de la même façon, mais ne conserve que les variables modifiées ou créées par le verbe.
5.5 La boîte à outils pour créer et modifier des variables avec R
5.5.1 Manipuler des variables numériques
Vous pouvez utiliser beaucoup de fonctions pour créer des variables avec mutate() :
les opérations arithmétiques :
+,-,*,/,^;arithmétique modulaire :
%/%(division entière) et%%(le reste), oùx == y * (x %/% y) + (x %% y);logarithmes :
log(),log2(),log10();navigations entre les lignes :
lead()etlag()qui permettent d’avoir accès à la valeur suivante et précédente d’une variable.
a <- data.frame(x=sample(1:10))
b <- mutate(a, lagx = lag(x),
leadx = lead(x),
lag2x = lag(x, n = 2),
lead2x = lead(x, n = 2))
datatable(b)opérations cumulatives ou glissantes :
R fournit des fonctions pour obtenir des opérations cumulatives les somme, produit, minimum et maximum cumulés, dplyr fournit l’équivalent pour les moyennes :
cumsum(),cumprod(),cummin(),cummax(),cummean()Pour appliquer des opérations glissantes, on peut soit créer l’opération avec l’instruction
lag(), soit exploiter le packageRcppRollqui permet d’exploiter des fonctions prédéfinies.
Exemple de somme glissante sur un pas de 2 observations.
a <- data.frame(x = sample(1:10))
b <- mutate(a, cumsumx = cumsum(x),
rollsumrx = roll_sumr(x, n = 2))
datatable(b)Attention aux différences entre roll_sum() et roll_sumr(). Contrairement à roll_sum(), la fonction roll_sumr() fait en sorte d’obtenir un vecteur de même dimension que l’entrée :
a$x## [1] 9 2 6 3 8 7 5 1 10 4rollsumrx <- roll_sumr(a$x, n=2)
rollsumx <- roll_sum(a$x, n=2)
length(rollsumrx) == length(a$x)## [1] TRUElength(rollsumx) == length(a$x)## [1] FALSEAussi dans le cadre d’opérations sur les dataframes, roll_sum() ne fonctionnera pas.
b <- mutate(a, cumsumx = cumsum(x),
rollsumx = roll_sum(x, n=2))Comparaisons logiques :
<,<=,>,>=,!=Rangs :
min_rank()devrait être la plus utile, il existe aussi notammentrow_number(),dense_rank(),percent_rank(),cume_dist(),ntile().coalesce(x, y): permet de remplacer les valeurs manquantes de x par celle de yvariable = ifelse(condition(x), valeursioui, valeursinon)permet d’affecter valeursi ou valeursinon à variable en fonction du fait que x répond à condition. Exemple : création d’une variable résultat pour savoir si les résultats de nos analyses sont bons, ou non.
analyseb <- mutate(analyse, resultat_ok = ifelse(code_remarque %in% c(1, 2, 7, 10),
yes = TRUE, no = FALSE))qui peut se résumer, lorsque yes = TRUE et no = FALSE, à :
analyseb <- mutate(analyse, resultat_ok = code_remarque %in% c(1, 2, 7, 10))case_when()permet d’étendre la logique deifelse()à des cas plus complexes. Les conditions mises dans uncase_when()ne sont pas exclusives. De ce fait, il faut pouvoir déterminer l’ordre d’évaluation des conditions qui y sont posées. Cet ordre s’effectue de bas en haut, c’est à dire que la dernière condition évaluée (celle qui primera sur toutes les autres) sera la première à écrire. Exemple: On va ici calculer des seuils fictifs sur les analyses.
analyseb <- mutate(analyse, classe_resultat_analyse = case_when(
resultat_analyse == 0 ~ "1",
resultat_analyse <= 0.001 ~ "2",
resultat_analyse <= 0.01 ~ "3",
resultat_analyse <= 0.1 ~ "4",
resultat_analyse > 0.1 ~ "5",
TRUE ~ ""
))5.5.2 Exercice 1 : Les données mensuelles sitadel
cf. package d’exercices {savoirfR}
À partir du fichier sitadel de février 2017 (ROES_201702.xls), produire un dataframe ‘sit_pdl_ind’ contenant pour la région Pays-de-la-Loire (code région 52), pour chaque mois et pour les logements individuels (définis par la somme des logements individuels purs et individuels groupés : i_AUT = ip_AUT + ig_AUT) :
- le cumul des autorisations sur 12 mois glissants (i_AUT_cum12),
- le taux d’évolution du cumul sur 12 mois (i_AUT_cum_evo, en %),
- la part de ce cumul dans celui de l’ensemble des logements autorisés (log_AUT), en pourcentage.
Résultat attendu :
solution sans le pipe (apercu des premières lignes) %>%
## # A tibble: 6 × 12
## date REG log_AUT ip_AUT ig_AUT colres_AUT i_AUT i_AUT_cum12
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 200001 52 1789 1266 245 278 1511 NA
## 2 200002 52 2022 1529 175 318 1704 NA
## 3 200003 52 2270 1466 205 599 1671 NA
## 4 200004 52 2040 1237 162 641 1399 NA
## 5 200005 52 2361 1357 357 647 1714 NA
## 6 200006 52 2504 1436 250 818 1686 NA
## # ℹ 4 more variables: i_AUT_cum12_lag12 <dbl>, i_AUT_cum_evo <dbl>,
## # log_AUT_cum12 <dbl>, part_i_AU <dbl>solution avec le pipe (apercu des premières lignes) %>%
## # A tibble: 6 × 12
## date REG log_AUT ip_AUT ig_AUT colres_AUT i_AUT i_AUT_cum12
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 200001 52 1789 1266 245 278 1511 NA
## 2 200002 52 2022 1529 175 318 1704 NA
## 3 200003 52 2270 1466 205 599 1671 NA
## 4 200004 52 2040 1237 162 641 1399 NA
## 5 200005 52 2361 1357 357 647 1714 NA
## 6 200006 52 2504 1436 250 818 1686 NA
## # ℹ 4 more variables: i_AUT_cum12_lag12 <dbl>, i_AUT_cum_evo <dbl>,
## # log_AUT_cum12 <dbl>, part_i_AU <dbl>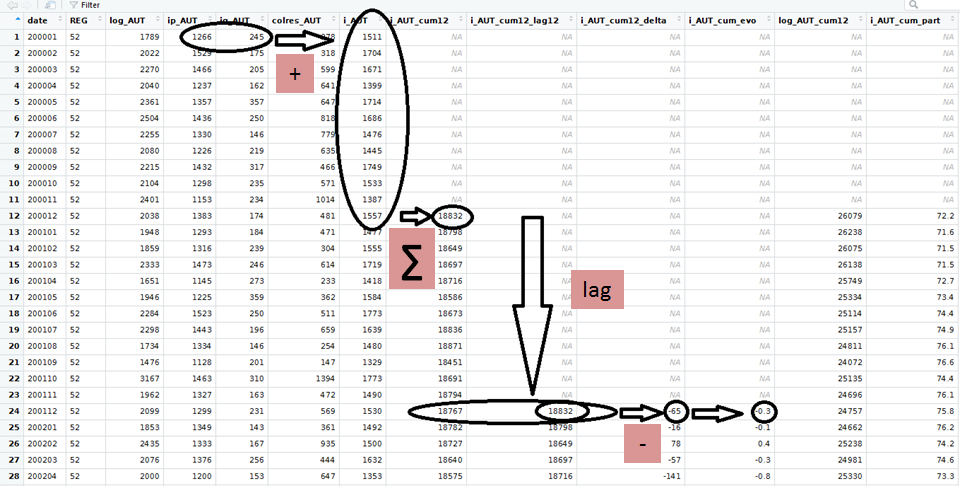
5.5.3 Manipuler des dates
Parmi l’ensemble des manipulations de variables, celle des dates et des heures est toujours une affaire complexe.
Le framework tidyverse propose le package {lubridate} qui permet de gérer ces informations de façon cohérente.
- gestion des dates :
dmy("jeudi 21 novembre 2020")
dmy("21112020")
ymd("20201121")- gestion des dates/heures :
dmy_hms("mardi 21 novembre 2020 9:30:00")
now()- combien de jours avant Noël ?
annee_en_cours <- year(today())
prochain_noel <- paste("25 décembre", annee_en_cours)
prochain_noel
dmy(prochain_noel) - today()- le jour de la semaine d’une date :
wday(dmy("19012038"), label = TRUE)Les fonctions make_date() et make_datetime() vous permettent de transformer un ensemble de variables en un format date ou date - heure. C’est par exemple utile lorsque l’on a des variables séparées pour l’année, le mois et le jour.
5.5.3.1 Exercice 2 : les dates
Convertir les colonnes de la table exercice au format date (quand c’est pertinent). La table exercice est issue de FormationPreparationDesDonnees.RData.
Résultat attendu :
## Rows: 153,497
## Columns: 22
## $ code_analyse <int> 5186581, 280131, 1576225, 799894, 472800, 27671…
## $ code_laboratoire <dbl> NA, 292, NA, NA, 292, NA, NA, NA, NA, NA, NA, N…
## $ code_prelevement <int> 37593, 7715, 15517, 9566, 8332, 26792, 35625, 1…
## $ code_parametre <dbl> 1216, 1668, 1185, 1217, 1907, 1945, 1673, 1234,…
## $ code_fraction_analysee <int> 23, 23, 23, 23, 23, 23, 23, 23, 23, 23, 23, 23,…
## $ resultat_analyse <dbl> 0.007, 0.050, 0.040, 0.050, 0.260, 0.020, 0.010…
## $ code_remarque <int> 10, 2, 2, 2, 1, 10, 10, 10, 10, 10, 10, 10, 2, …
## $ limite_detection <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
## $ limite_quantification <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
## $ code_intervenant <fct> NA, 104, NA, NA, 104, NA, NA, 53, NA, 44, 49, 4…
## $ code_reseau <fct> OSUR, OSUR, FREDON, OSUR, OSUR, OSUR, OSUR, ARS…
## $ code_station <chr> "04153800", "04130000", "04132500", "04214000",…
## $ date_prelevement <date> 2014-09-16, 2003-08-05, 2008-09-01, 2007-05-02…
## $ code_support <int> NA, 3, NA, NA, 3, NA, NA, 3, NA, 3, 3, 3, NA, N…
## $ libelle_station <chr> "MOZEE à CHANTONNAY", "MAYENNE à DAON", "MAYENN…
## $ date_creation <date> 1900-01-01, 1900-01-01, 1900-01-01, 1900-01-01…
## $ source <chr> "AELB", "AELB", "AELB", "AELB", "AELB", "AELB",…
## $ code_masse_eau <chr> "GR1950", "GR0460c", "GR0460c", "GR0121", "GR04…
## $ code_entite_hydro <chr> "N3036200", "M---0090", "M---0090", "J78-0300",…
## $ code_troncon_hydro <chr> "N3036200", "M3620090", "M3910090", "J7800300",…
## $ code_commune <chr> "85051", "53089", "49214", "44036", "53017", "5…
## $ date_formatee <chr> "16/09/2014", "05/08/2003", "01/09/2008", "02/0…5.5.4 Manipuler des chaînes de caractères
Le package {stringr} compile l’ensemble des fonctions de manipulation de chaînes de caractère utiles sur ce type de données.
On peut diviser les manipulations de chaînes de caractères en 4 catégories :
- manipulations des caractères eux-mêmes,
- gestion des espaces,
- opérations liées à la langue,
- manipulations de “pattern”, notamment des expressions régulières.
5.5.4.1 Manipulations sur les caractères
Obtenir la longueur d’une chaîne avec str_length() :
library(stringr)
str_length("abc")## [1] 3Extraire une chaîne de caractères avec str_sub()
str_sub() prend 3 arguments : une chaîne de caractère, une position de début, une position de fin.
Les positions peuvent être positives, et dans ce cas, on compte à partir de la gauche, ou négatives, et dans ce cas on compte à partir de la droite.
a <- data.frame(x = c(" libeatg", "delivo y"))
b <- mutate(a, pos3a4 = str_sub(string = x, start = 3, end = 4),
pos3a2avtlafin = str_sub(string = x, start = 3, end = -2))
datatable(b)str_sub() peut être utilisé pour remplacer un caractère
str_sub(a$x, start = 6, end = 9) <-"rer"
a$x## [1] " liberer" "delivrer"Si on souhaite réaliser ce genre d’opération dans le cadre d’un mutate, il faut utiliser
une fonction dite “pipe-operator-friendly”, par exemple stri_sub_replace() du package {stringi}
# install.packages("stringi")
library(stringi)
a <- data.frame(x = c(" libeatg", "delivo y"))
b <- mutate(a, y=stri_sub_replace(str=x, from=6, to=9, value = "rer"))
datatable(b)5.5.4.2 Gestion des espaces
La fonction str_pad() permet de compléter une chaîne de caractère pour qu’elle atteigne une taille fixe. Le cas typique d’usage est la gestion des codes communes Insee.
code_insee <- 1001
str_pad(code_insee, 5, pad = "0")## [1] "01001"On peut choisir de compléter à gauche, à droite, et on peut choisir le “pad”. Par défaut, celui-ci est l’espace.
La fonction inverse de str_pad() est str_trim() qui permet de supprimer les espaces aux extrémités de notre chaîne de caractères.
proust <- " Les paradoxes d'aujourd'hui sont les préjugés de demain. "
str_trim(proust)## [1] "Les paradoxes d'aujourd'hui sont les préjugés de demain."str_trim(proust, side = "left")## [1] "Les paradoxes d'aujourd'hui sont les préjugés de demain. "Les expressions régulières permettent la détection de “patterns” sur des chaînes de caractères. Par exemple “^” sert à indiquer que la chaîne de caractère recherchée doit se trouver au début de la chaîne examinée. Au contraire, “$” sert à indiquer que la chaîne de caractère recherchée doit se trouver à la fin.
a <- data.frame(txt = c("vélo", "train", "voilier", "bus", "avion", "tram", "trottinette"))
b <- mutate(a, tr_au_debut = str_detect(string = txt, pattern = "^tr"))
b## txt tr_au_debut
## 1 vélo FALSE
## 2 train TRUE
## 3 voilier FALSE
## 4 bus FALSE
## 5 avion FALSE
## 6 tram TRUE
## 7 trottinette TRUEfilter(b, tr_au_debut)## txt tr_au_debut
## 1 train TRUE
## 2 tram TRUE
## 3 trottinette TRUEfilter(a, str_detect(string = txt, pattern = "n$"))## txt
## 1 train
## 2 avion5.5.4.3 Opérations liées à la langue
Ces différentes fonctions ne donneront pas le même résultat en fonction de la langue par défaut utilisée. La gestion des majuscules/minuscules :
proust <- "Les paradoxes d'aujourd'hui sont LES préjugés de Demain."
str_to_upper(proust)## [1] "LES PARADOXES D'AUJOURD'HUI SONT LES PRÉJUGÉS DE DEMAIN."str_to_lower(proust)## [1] "les paradoxes d'aujourd'hui sont les préjugés de demain."str_to_title(proust)## [1] "Les Paradoxes D'aujourd'hui Sont Les Préjugés De Demain."La gestion de l’ordre, str_sort() et str_order() :
a <- data.frame(x = c("y", "i", "k"))
mutate(a, en_ordre = str_sort(x),
selon_position = str_order(x))## x en_ordre selon_position
## 1 y i 2
## 2 i k 3
## 3 k y 1Suppression des accents (base::iconv) :
proust2 <- "Les paradoxes d'aujourd'hui sont les préjugés de demain ; et ça c'est embêtant"
iconv(proust2, to = "ASCII//TRANSLIT")## [1] "Les paradoxes d'aujourd'hui sont les prejuges de demain ; et ca c'est embetant"Avec humour, un petit aide-mémoire illustré, très visuel, est proposé par Lise Vaudor ici.
5.5.5 Manipuler des variables factorielles ( = qualitatives ou catégorielles)
Les facteurs (ou factors, an anglais) sont un type de vecteur géré nativement par R qui permettent de gérer les variables qualitatives ou catégorielles. Les facteurs sont souvent mis en regard des données labellisées utilisées dans d’autres logiciels statistiques. Les facteurs possèdent un attribut appelé niveaux (levels, en anglais) qui contient l’ensemble des valeurs qui peuvent être prises par les éléments du vecteur.
Les fonctions du module {forcats} permettent de modifier les modalités d’une variable factorielle, notamment :
changer les modalités des facteurs et/ou leur ordre,
regrouper des modalités.
On va ici utiliser la fonction fct_infreq(), pour modifier le tri des stations en fonction de leur fréquence d’apparition dans la table “prelevement”.
{forcats} permet beaucoup d’autres possibilités de tri :
tri manuel des facteurs avec
fct_relevel();en fonction de la valeur d’une autre variable avec
fct_reorder();en fonction de l’ordre d’apparition des modalités avec
fct_inorder().
Consulter la documentation du package {forcats} pour voir toutes les possibilités très riches de ce module.
En quoi ces fonctions sont utiles ?
Elles permettent notamment :
lorsqu’on fait des graphiques, d’afficher les occurences les plus importantes d’abord ;
de lier l’ordre d’une variable en fonction d’une autre (par exemple les code Insee des communes en fonction des régions).
Exemple : ordonner les modalités d’un facteur pour améliorer l’aspect d’un graphique
library(ggplot2)
library(forcats)
data <- data.frame(num = c(1, 8, 4, 3, 6, 7, 5, 2, 11, 3),
cat = c(letters[1:10]))
ggplot(data, aes(x = cat, num)) +
geom_bar(stat = "identity") +
xlab(label = "Facteur") + ylab(label = "Valeur")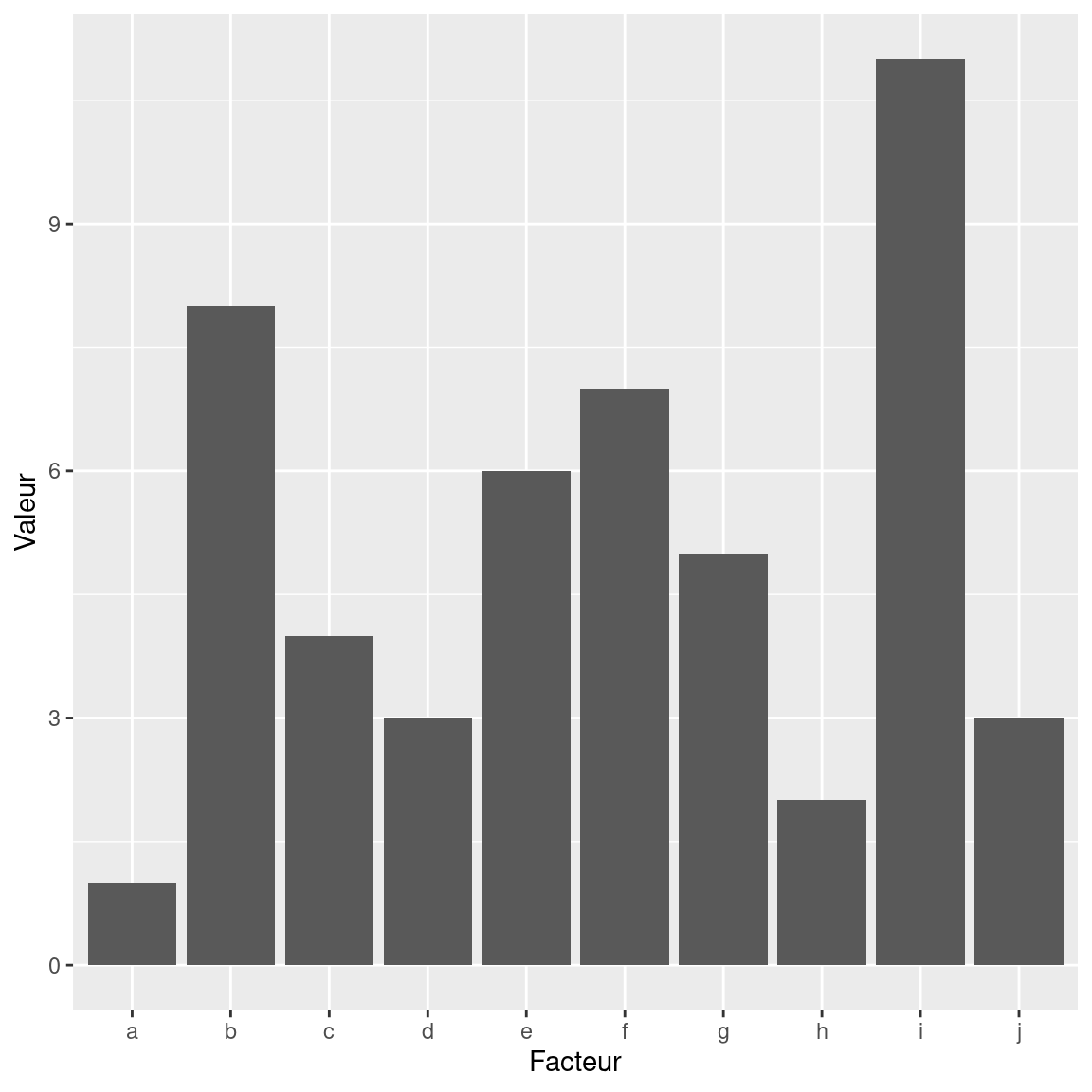
ggplot(data, aes(x = fct_reorder(cat, -num), num)) +
geom_bar (stat = "identity") +
xlab(label = "Facteur ordonné") + ylab(label = "Valeur")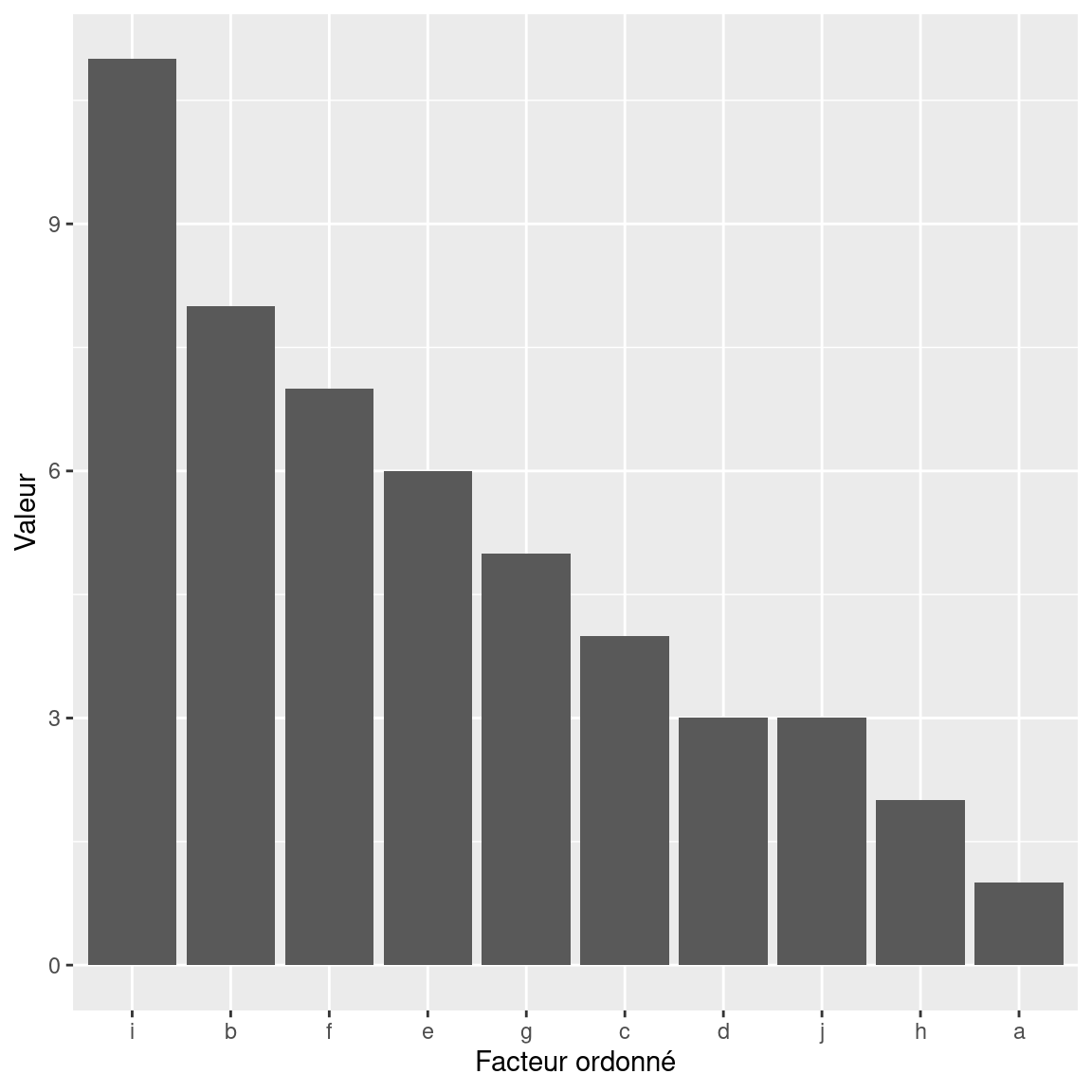
5.6 Agréger des données : summarise()
La fonction summarise() permet d’agréger des données, en appliquant une fonction sur les variables pour construire une statistique sur les observations de la table.
summarise() est une fonction dite de “résumé”. À l’inverse de mutate(), quand une fonction summarise est appelée, elle retourne une seule information. La moyenne, la variance, l’effectif… sont des informations qui condensent la variable étudiée en une seule information.
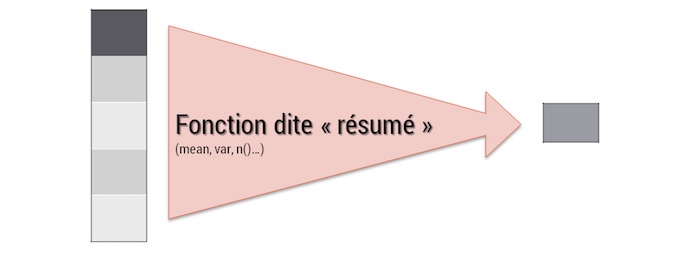
La syntaxe de summarise est classique. Le résultat est un dataframe.
summarise(exercice,
mesure_moyenne = mean(resultat_analyse, na.rm = TRUE))On peut calculer plusieurs statistiques sur une agrégation
summarise(exercice,
mesure_moyenne = mean(resultat_analyse, na.rm = TRUE),
mesure_total = sum(resultat_analyse, na.rm = TRUE)
)5.6.1 Quelques fonctions d’agrégations utiles
- compter :
n() - sommer :
sum() - compter des valeurs non manquantes
sum(!is.na()) - moyenne :
mean(), moyenne pondérée :weighted.mean() - écart-type :
sd() - médiane :
median(), quantile :quantile(.,quantile) - minimum :
min(), maximum :max() - position :
first(),nth(., position),last()
La plupart de ces fonctions d’agrégation sont paramétrables pour indiquer comment traiter les valeurs manquantes (NA) grâce à l’argument na.rm.
Si on ne souhaite pas tenir compte des valeurs manquantes pour effectuer notre synthèse, il faut indiquer na.rm = TRUE pour évacuer les valeurs manquantes du calcul, sinon, le résultat apparaîtra comme lui même manquant, car il manque des observations pour pouvoir calculer correctement notre résultat.
C’est la connaissance de votre source de données et du travail en court qui déterminera comment vous souhaitez que les valeurs manquantes soit traitées.
5.7 Agréger des données par dimension : group_by()
La fonction summarise() est utile, mais la plupart du temps, nous avons besoin non pas d’agréger des données d’une table entière, mais de construire des agrégations sur des sous-ensembles : par année, département…
La fonction group_by() va permettre d’éclater notre table en fonction de dimensions de celle-ci.
Ainsi, si on veut construire des statistiques agrégées non sur l’ensemble de la table, mais pour chacune des modalités d’une ou de plusieurs variables de la table. Il faut deux étapes :
utiliser préalablement la fonction
group_by()pour définir la ou les variables sur lesquelles on souhaite agréger les données,utiliser
summarise()sur la table en sortie de l’étape précédente.
Découper un jeu de données pour réaliser des opérations sur chacun des sous-ensembles afin de les restituer ensuite de façon organisée est appelée stratégie du split – apply – combine schématiquement, c’est cette opération qui est réalisée par dplyr dès qu’un group_by() est introduit sur une table.
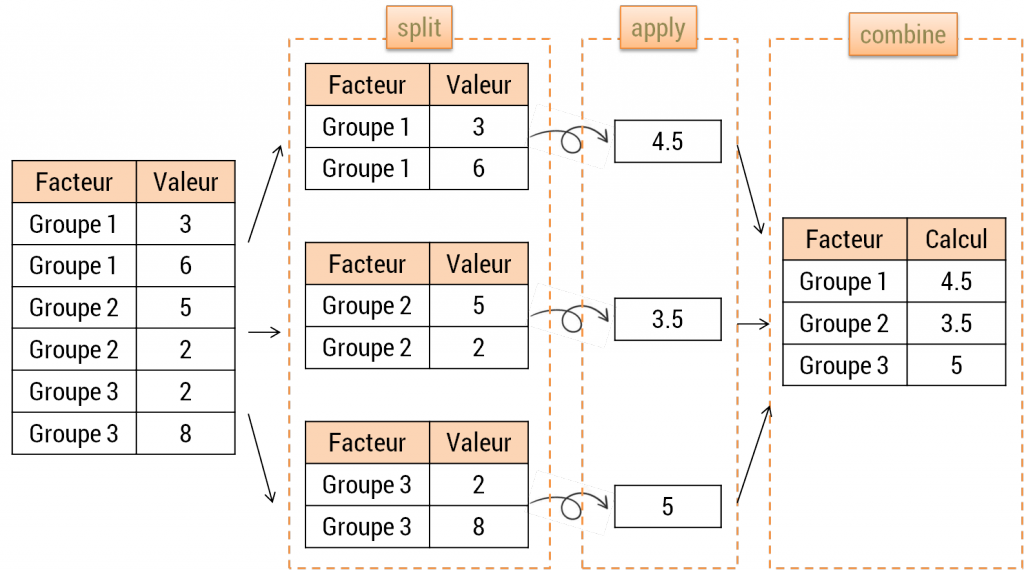
Exemple pour calculer les statistiques précédentes par année :
exercice <- mutate(exercice, annee = year(date_prelevement))
paran <- group_by(exercice, annee)
summarise(paran,
mesure_moyenne = mean(resultat_analyse, na.rm = TRUE),
mesure_total = sum(resultat_analyse, na.rm = TRUE))## # A tibble: 26 × 3
## annee mesure_moyenne mesure_total
## <dbl> <dbl> <dbl>
## 1 1991 0.0724 1.38
## 2 1992 0.192 4.42
## 3 1993 0.137 2.46
## 4 1994 0.07 2.24
## 5 1995 0.0687 2.06
## 6 1996 0.0867 3.99
## 7 1997 0.0520 2.50
## 8 1998 0.145 22.8
## 9 1999 0.0672 44.6
## 10 2000 0.0586 36.9
## # ℹ 16 more rowsPour reprendre des traitements “table entière”, il faut mettre fin au group_by() par un ungroup().
La fonction summarise() accepte désormais un argument .groups qui permet d’indiquer directement comment nous souhaitons voir ré-assemblé ou non notre jeu de données.
paran <- group_by(exercice, annee, code_reseau)
resultat <- summarise(paran, mesure_moyenne = mean(resultat_analyse, na.rm = TRUE),
mesure_total = sum(resultat_analyse, na.rm = TRUE))## `summarise()` has grouped output by 'annee'. You can override using the
## `.groups` argument.Si on omet de lui déclarer comment traiter les groupes en sortie, summarise() nous informe des éventuels groupes résiduels, ici resultat est toujours groupé par annee.
Pour remédier à ce message ou changer le comportement de summarise(), .groups peut prendre plusieurs valeurs :
"drop_last": va supprimer le dernier niveau de groupement de notre jeu de données. Dans notre exemple le groupe seloncode_reseauva disparaître et celui lié àanneeva rester. C’est le comportement par défaut."drop": supprime tous les niveaux de groupement"keep": conserve tous les niveaux de groupement."rowwise": chaque ligne devient son propre groupe.
resultat <- summarise(paran, mesure_moyenne = mean(resultat_analyse, na.rm = TRUE), mesure_total = sum(resultat_analyse, na.rm = TRUE),
.groups = "drop")5.8 Le pipe

Le pipe est la fonction qui va vous permettre d’écrire votre code de façon plus lisible pour vous et les utilisateurs.
Comment ?
En se rapprochant de l’usage usuel en grammaire.
verbe(sujet, complement) devient sujet %>% verbe(complement)
Quand on enchaîne plusieurs verbes, l’avantage devient encore plus évident :
verbe2(verbe1(sujet, complement1), complement2) devient sujet %>% verbe1(complement1) %>% verbe2(complement2)
En reprenant l’exemple précédent, sans passer par les étapes intermédiaires, le code aurait cette tête :
summarise (
group_by (
mutate (
exercice,
annee = year(date_prelevement)
),
annee
),
mesure_moyenne = mean(resultat_analyse, na.rm = TRUE),
mesure_total = sum(resultat_analyse, na.rm = TRUE)
)## # A tibble: 26 × 3
## annee mesure_moyenne mesure_total
## <dbl> <dbl> <dbl>
## 1 1991 0.0724 1.38
## 2 1992 0.192 4.42
## 3 1993 0.137 2.46
## 4 1994 0.07 2.24
## 5 1995 0.0687 2.06
## 6 1996 0.0867 3.99
## 7 1997 0.0520 2.50
## 8 1998 0.145 22.8
## 9 1999 0.0672 44.6
## 10 2000 0.0586 36.9
## # ℹ 16 more rowsAvec l’utilisation du pipe (raccourci clavier CTrl + Maj + M), il devient :
exercice %>%
mutate(annee = year(date_prelevement)) %>%
group_by(annee) %>%
summarise(mesure_moyenne = mean(resultat_analyse, na.rm = TRUE),
mesure_total = sum(resultat_analyse, na.rm = TRUE))## # A tibble: 26 × 3
## annee mesure_moyenne mesure_total
## <dbl> <dbl> <dbl>
## 1 1991 0.0724 1.38
## 2 1992 0.192 4.42
## 3 1993 0.137 2.46
## 4 1994 0.07 2.24
## 5 1995 0.0687 2.06
## 6 1996 0.0867 3.99
## 7 1997 0.0520 2.50
## 8 1998 0.145 22.8
## 9 1999 0.0672 44.6
## 10 2000 0.0586 36.9
## # ℹ 16 more rows5.9 La magie des opérations groupées
L’opération group_by() que nous venons de voir est très utile pour les agrégations, mais elle peut aussi servir pour créer des variables ou filtrer une table, puisque group_by() permet de traiter notre table en entrée comme autant de tables séparées par les modalités des variables de regroupement.
5.9.1 Exercice 3
A partir des données “sitadel” chargées dans l’exercice 1, effectuer les opérations suivantes en utilisant l’opérateur %>% :
- effectuer les mêmes calculs que ceux réalisés sur la région 52, mais sur chacune des régions –> à stocker dans ‘sit_ind’
- calculer les agrégations par année civile pour chacune des régions, puis leur taux d’évolution d’une année sur l’autre (exemple : (val2015-val2014)/val2014) –> à stocker dans ‘sit_annuel’
Résultat attendu pour sit_ind :
## # A tibble: 5,356 × 12
## date REG log_AUT ip_AUT ig_AUT colres_AUT i_AUT i_AUT_cum12
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 200001 01 440 194 12 234 206 NA
## 2 200001 02 372 189 14 169 203 NA
## 3 200001 03 172 25 3 144 28 NA
## 4 200001 04 473 325 84 64 409 NA
## 5 200001 11 3029 754 318 1957 1072 NA
## 6 200001 21 547 274 94 179 368 NA
## 7 200001 22 475 328 16 131 344 NA
## 8 200001 23 569 445 35 89 480 NA
## 9 200001 24 1057 714 88 255 802 NA
## 10 200001 25 708 410 206 92 616 NA
## # ℹ 5,346 more rows
## # ℹ 4 more variables: i_AUT_cum12_lag12 <dbl>, i_AUT_cum_evo <dbl>,
## # log_AUT_cum12 <dbl>, part_i_AU <dbl>Résultat attendu pour sit_annuel :
## # A tibble: 468 × 10
## REG annee log_AUT ip_AUT ig_AUT colres_AUT evol_an_log_AUT evol_an_ip_AUT
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 01 2000 6625 2776 674 3175 NA NA
## 2 02 2000 3956 1805 270 1881 NA NA
## 3 03 2000 1501 363 363 775 NA NA
## 4 04 2000 9749 4580 1246 3923 NA NA
## 5 11 2000 44443 8843 4836 30764 NA NA
## 6 21 2000 5519 3164 890 1465 NA NA
## 7 22 2000 6363 3819 721 1823 NA NA
## 8 23 2000 8803 4712 1256 2835 NA NA
## 9 24 2000 13386 7770 1867 3749 NA NA
## 10 25 2000 8678 5288 1401 1989 NA NA
## # ℹ 458 more rows
## # ℹ 2 more variables: evol_an_ig_AUT <dbl>, evol_an_colres_AUT <dbl>5.9.2 Exercice 4
Sur les données FormationPreparationDesDonnees.RData, table exercice :
1/ calculer le taux de quantification pour chaque molécule et chacune des années : chaque molécule est identifiée par son code_parametre,
le taux de quantification est le nombre de fois qu’une molécule est retrouvée (càd si code_remarque = 1) sur le nombre de fois où elle a été
cherchée (càd si code_remarque = 1, 2, 7 ou 10). Pour cela :
- créer la variable
annee - créer la variable de comptage des présences pour chaque analyse (1=présent, 0=absent)
- créer la variable de comptage des recherches pour chaque analyse (1=recherchée, 0=non recherchée)
- pour chaque combinaison
anneexcode_parametre, calculer le taux de quantification
2/ trouver pour chaque station, sur l’année 2016, le prélèvement pour lequel la concentration cumulée, toutes substances confondues, est la plus élevée (~ le prélèvement le plus pollué). Pour cela :
- filtrer les concentrations quantifiées (
code_remarque=1) et l’année 2016 - sommer les concentrations (
resultat_analyse) par combinaisoncode_stationxcode_prelevement - ne conserver que le prélèvement avec le concentration maximale
Résultats attendus :
Résultat attendu pour le taux de quantification par molécule et année :
## # A tibble: 6,538 × 3
## annee code_parametre taux_quantif
## <dbl> <dbl> <dbl>
## 1 1991 1129 0
## 2 1991 1130 0
## 3 1991 1176 0
## 4 1991 1199 0
## 5 1991 1212 0
## 6 1991 1259 0
## 7 1991 1263 100
## 8 1991 1267 0
## 9 1992 1101 0
## 10 1992 1107 100
## # ℹ 6,528 more rowsRésultat attendu pour prélèvement le plus pollué de chaque station en 2016 :
## # A tibble: 176 × 3
## libelle_station code_prelevement concentration_cumulee
## <chr> <int> <dbl>
## 1 ANGLE GUIGNARD-RETENUE 43003 0.04
## 2 ANXURE À SAINT-GERMAIN-D'ANXURE 42228 0.02
## 3 APREMONT-RETENUE 42895 0.035
## 4 ARAIZE à CHATELAIS 41451 0.006
## 5 ARON à MOULAY 41359 0.008
## 6 AUBANCE À LOUERRE 41571 0.08
## 7 AUBANCE à MURS-ERIGNE 41542 0.317
## 8 AUBANCE à SAINT-SATURNIN-SUR-LOIRE 41584 0.167
## 9 AUTHION à LES PONTS-DE-CE 42532 0.27
## 10 AUTISE À SAINT-HILAIRE-DES-LOGES 41998 0.048
## # ℹ 166 more rows5.10 Les armes non conventionnelles de la préparation des donnéees
Nous venons de voir les principaux verbes de manipulation d’une table de dplyr. Ces verbes acquièrent encore plus de puissance quand ils sont appelés avec les fonctions across() et/ou where().
5.10.1 Les select helpers
Répéter des opérations de nettoyage ou de typage sur les différentes variables d’un jeu de données peut s’avérer fastidieux lorsque l’on a à écrire les opérations variable par variable.
La fonction select() propose cinq manières différentes de désigner les variables à sélectionner.
Nous avons vu la première et la plus intuitive, qui est de nommer les variables une à une.
On peut également utiliser les : qui permettent de sélectionner une liste de variables consécutives.
On peut également désigner les variables à sélectionner en fonction de leur position :
select(exercice, code_analyse, code_laboratoire, code_prelevement, code_parametre,
code_fraction_analysee, resultat_analyse, code_remarque) %>%
names()## [1] "code_analyse" "code_laboratoire" "code_prelevement"
## [4] "code_parametre" "code_fraction_analysee" "resultat_analyse"
## [7] "code_remarque"select(exercice, code_analyse:code_remarque) %>% names()## [1] "code_analyse" "code_laboratoire" "code_prelevement"
## [4] "code_parametre" "code_fraction_analysee" "resultat_analyse"
## [7] "code_remarque"select(exercice, -c(code_analyse:code_remarque)) %>% names()## [1] "limite_detection" "limite_quantification" "code_intervenant"
## [4] "code_reseau" "code_station" "date_prelevement"
## [7] "code_support" "libelle_station" "date_creation"
## [10] "source" "code_masse_eau" "code_entite_hydro"
## [13] "code_troncon_hydro" "code_commune"select(exercice, 1:7) %>% names()## [1] "code_analyse" "code_laboratoire" "code_prelevement"
## [4] "code_parametre" "code_fraction_analysee" "resultat_analyse"
## [7] "code_remarque"select(exercice, -c(1:7)) %>% names()## [1] "limite_detection" "limite_quantification" "code_intervenant"
## [4] "code_reseau" "code_station" "date_prelevement"
## [7] "code_support" "libelle_station" "date_creation"
## [10] "source" "code_masse_eau" "code_entite_hydro"
## [13] "code_troncon_hydro" "code_commune"Sélectionner les variables en fonction de leur position peut sembler séduisant, mais attention aux problèmes de reproductibilité que cela peut poser si le jeu de données en entrée bouge un peu entre deux millésimes.
On peut également sélectionner des variables selon des conditions sur leur nom. Par exemple, on peut sélectionner les variables dont le nom commence par “date”, ou se termine par “station”, ou contient “prel” ou en fonction d’une expression régulière comme “m.n” (le nom contient un “m” suivi d’un caractère suivi d’un “n”.
select(exercice, starts_with("date")) %>% names()## [1] "date_prelevement" "date_creation"select(exercice, ends_with("station")) %>% names()## [1] "code_station" "libelle_station"select(exercice, contains("prel")) %>% names()## [1] "code_prelevement" "date_prelevement"select(exercice, matches("m.n")) %>% names()## [1] "code_prelevement" "date_prelevement" "code_commune"On peut également sélectionner des variables selon des conditions sur leur type, avec la fonction where(). Par exemple, sélectionner toutes les variables numériques ou toutes les variables de type caractère.
select(exercice, where(is.numeric)) %>% names()## [1] "code_analyse" "code_laboratoire" "code_prelevement"
## [4] "code_parametre" "code_fraction_analysee" "resultat_analyse"
## [7] "code_remarque" "limite_detection" "limite_quantification"
## [10] "code_support"select(exercice, where(is.character)) %>% names()## [1] "code_station" "date_prelevement" "libelle_station"
## [4] "date_creation" "source" "code_masse_eau"
## [7] "code_entite_hydro" "code_troncon_hydro" "code_commune"On peut enfin sélectionner des variables en combinant les moyens détaillés ci-avant et en recourant aux opérateurs booléens : ! (négation), & (et), | (ou).
select(exercice, 1:7 & starts_with("code")) %>% names()## [1] "code_analyse" "code_laboratoire" "code_prelevement"
## [4] "code_parametre" "code_fraction_analysee" "code_remarque"select(exercice, starts_with("date") & !where(is.Date)) %>% names()## [1] "date_prelevement" "date_creation"5.10.2 Utiliser les select helpers avec les autres verbes du tidyverse
5.10.2.1 rename() et rename_with()
Lorsqu’on souhaite renommer les variable une à une, la fonction rename() fonctionne de la même manière que select() :
mon_df_renomme <- rename(mon_dataframe, nouveau_nom1 = ancien_nom1, nouveau_nom2 = ancien_nom2)
Si l’on souhaite recourir aux select helpers, il faut utiliser rename_with(), avec la syntaxe rename_with(.data= mon_df, .fn= ma_fonction_de_renommage, .cols= les_variables_a_renommer). Exemple avec la fonction toupper() qui passe les chaînes de caractères en majuscules.
rename_with(station, toupper, starts_with("code")) %>% names()## [1] "CODE_STATION" "libelle_station" "date_creation"
## [4] "source" "CODE_MASSE_EAU" "CODE_ENTITE_HYDRO"
## [7] "CODE_TRONCON_HYDRO" "CODE_COMMUNE"Si la fonction de renommage est plus complexe qu’un simple mot, il faut recourir au pronom .x et au ~ pour la définir. Exemple avec la fonction str_sub() de {stringr} vue précédemment :
rename_with(exercice, ~ str_sub(.x, start = 6, end = str_length(.x)),
starts_with("code")) %>% names()## [1] "analyse" "laboratoire" "prelevement"
## [4] "parametre" "fraction_analysee" "resultat_analyse"
## [7] "remarque" "limite_detection" "limite_quantification"
## [10] "intervenant" "reseau" "station"
## [13] "date_prelevement" "support" "libelle_station"
## [16] "date_creation" "source" "masse_eau"
## [19] "entite_hydro" "troncon_hydro" "commune"5.10.3 filter(), mutate(), group_by(), summarise(), arrange(), transmute()…
Les autres verbes de {dplyr} ont besoin de la fonction across() pour fonctionner avec les select helpers. Comme pour rename_with(), les fonctions complexes sont à déclarer avec le ~ et le pronom .x. On peut en désigner plusieurs ou leur fournir un nom qui servira de suffixe aux noms des variables calculées, en passant la ou les fonctions dans une liste : .fn=list(suffixe1 = ma_fonction1, suffixe2 = ma_fonction2).
La syntaxe générale devient :
monverbe(.data, across(mesvariables, malistedefonctions),
across(mesvariables2, malistedefonctions2))filter(parametre, across(starts_with("date"), ~ .x > "2015-01-01")) %>%
select(1:7)## code_parametre nom_parametre statut_parametre
## 1 7782 Desméthyl-chlortoluron Validé
## 2 7801 Cyprosulfamide Validé
## 3 7783 Haloxyfop méthyl Validé
## 4 7748 cyflufénamide Validé
## date_creation_parametre date_maj_parametre auteur_parametre parametre_calcule
## 1 2015-03-10 2015-03-27 INOVALYS Nantes FALSE
## 2 2015-04-30 2015-06-10 AERM FALSE
## 3 2015-03-10 2015-03-27 INOVALYS FALSE
## 4 2015-02-13 2015-02-13 CARSO-LSEHL FALSEmutate(exercice, across(starts_with("code") & where(is.numeric), as.factor),
across(starts_with("date"), as.Date)) %>%
head() %>%
datatable()summarise(parametre, across(starts_with("code"), n_distinct))## code_parametre
## 1 435group_by(prelevement, across(code_intervenant:code_station)) %>%
summarise(across(everything(), list(nb = n_distinct)), .groups = "drop")## # A tibble: 766 × 6
## code_intervenant code_reseau code_station code_prelevement_nb
## <fct> <fct> <chr> <int>
## 1 44 ARS 044000001 51
## 2 44 ARS 044000044 7
## 3 44 ARS 044000045 5
## 4 44 ARS 044000046 4
## 5 44 ARS 044000047 3
## 6 44 ARS 044000048 4
## 7 44 ARS 044000070 6
## 8 44 ARS 044000071 5
## 9 44 ARS 044000076 4
## 10 44 ARS 044000077 5
## # ℹ 756 more rows
## # ℹ 2 more variables: date_prelevement_nb <int>, code_support_nb <int>Exemple sur l’exercice sur les données sitadel.
sitadel <- read_excel("extdata/ROES_201702.xls", "AUT_REG") %>%
group_by(REG) %>%
mutate(across(where(is.numeric), list(cumul12 = ~ roll_sumr(.x, n = 12))),
across(ends_with("cumul12"), list(evo = ~ 100 * .x / lag (.x, 12) - 100,
part = ~ 100 *.x / log_AUT_cumul12)))
datatable(sitadel)