Chapitre 6 Manipuler plusieurs tables
6.1 Concepts généraux
Le package {dplyr} possède également plusieurs fonctions permettant de travailler sur deux tables.
On va pouvoir regrouper ces fonctions en plusieurs catégories de manipulations :
- pour fusionner des informations de deux tables entre elles : jointures transformantes,
- pour sélectionner des observations d’une table en fonction de celles présentes dans une autre table : jointures filtrantes,
- pour traiter deux tables ayant les mêmes colonnes et sélectionner sur celles-ci des observations de l’une et l’autre : opérations ensemblistes,
- des manipulations visant à additionner deux tables ensembles : assemblages.
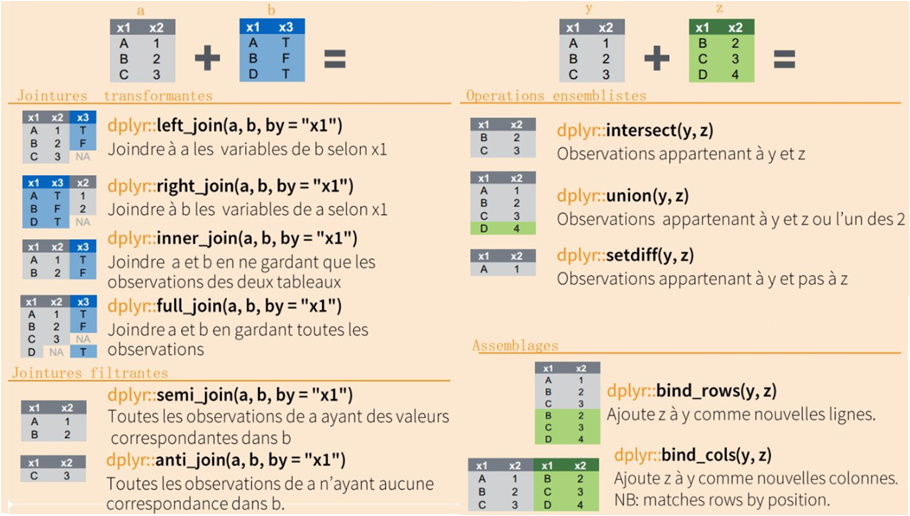
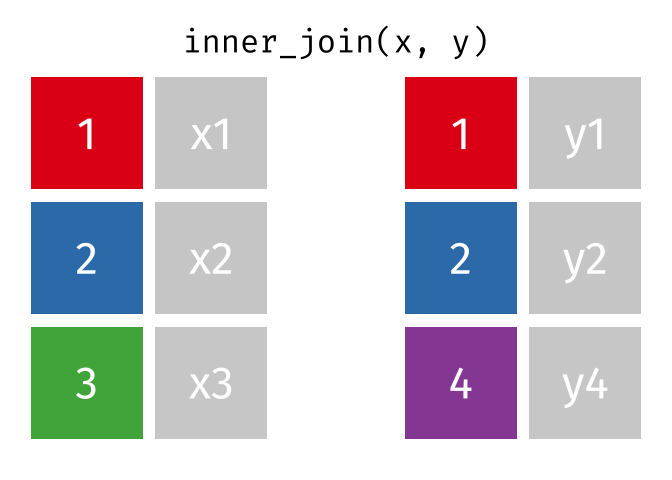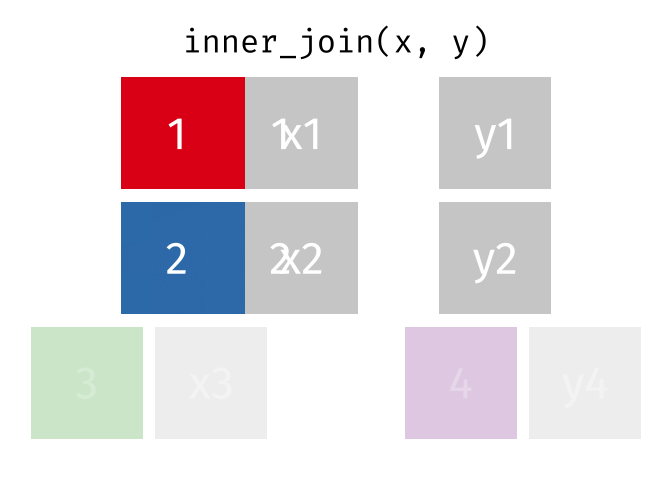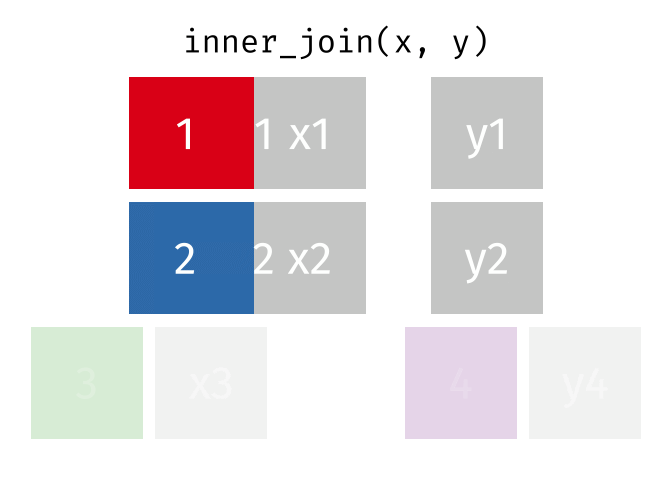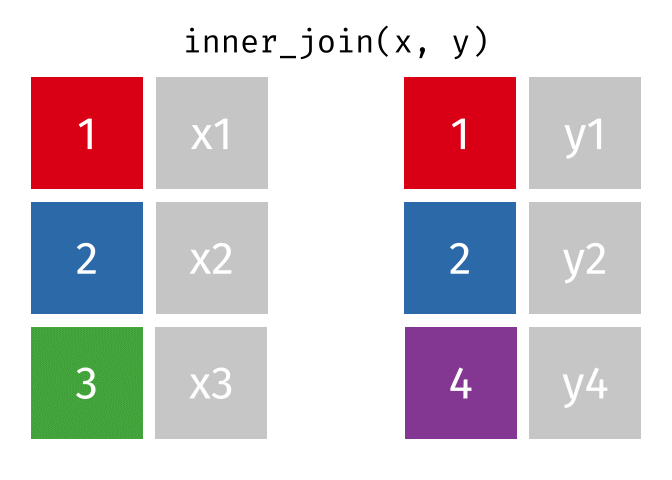
6.2 Principaux exemples de jointure
Dans la capture ci-dessus, les opérations de jointure supposent que la colonne x1 est présente dans a et b.
Voici quelques exemples détaillant les principaux cas rencontrés :
# jointure avec des noms de colonnes différentes pour la clé
# `colonne_c` est une colonne de data_a et `colonne_d` est une colonne de data_b
left_join(data_a, data_b, by = c("colonne_c" = "colonne_d"))
# jointure sur deux colonnes
# `colonne_e` et `colonne_f` sont des colonnes de data_a et de data_b
left_join(data_a, data_b, by = c("colonne_e", "colonne_f"))
# jointure sur deux colonnes avec noms différents
# `colonne_g`et `colonne_i` sont des colonnes de data_a, `colonne_h` et `colonne_j` sont des colonnes de data_b
left_join(data_a, data_b, by = c("colonne_g" = "colonne_h", "colonne_i" = "colonne_j"))6.3 Paramétrage du suffixe pour des colonnes présentes dans les deux tables
Dans le cas où des colonnes (hors jointure) sont présentes dans data_a et data_b, ces colonnes seront suffixées par :
.xpour les colonnes provenant dedata_a.ypour les colonnes provenant dedata_b
Il est possible de modifier ces suffixes en passant le paramètre suffix = c("_a", "_b") au moment de la jointure.
left_join(data_a, data_b, by = c("colonne_c" = "colonne_d"), suffix = c("_a", "_b"))6.4 Animations de Garrick Abenduie
Les animations proviennent de : https://www.garrickadenbuie.com/project/tidyexplain/
6.5 Exercice 5
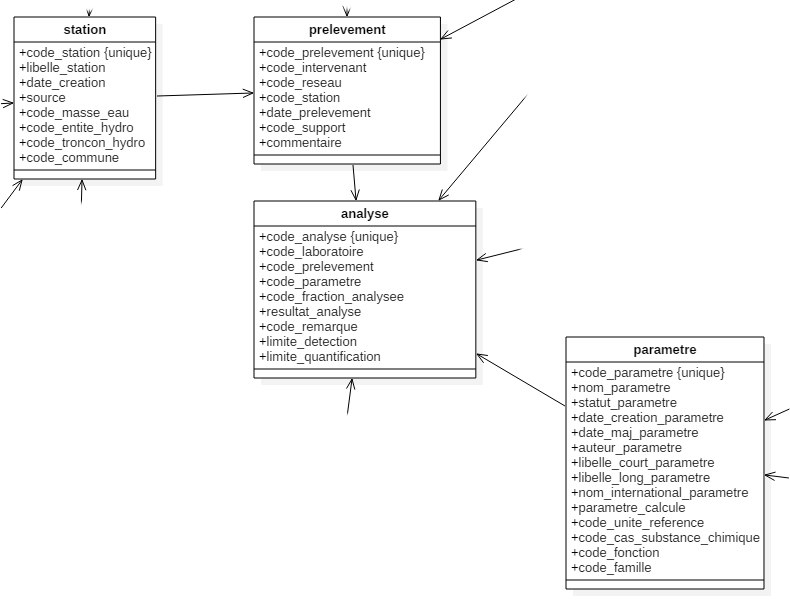
Dans les données FormationPreparationDesDonnees.RData, chaque analyse est effectuée à partir d’un prélèvement, lui-même réalisé au niveau d’une station.
Reconstituer le dataframe
exercicequi rassemble les données contenues dans les tablesanalyse,prelevementetstationgrâce à des jointures. Il faut partir de la tableanalyseet faire des jointurescalculer le nombre d’analyses réalisées sur des molécules (identifiables par leur
code_parametre) et décrites dans le référentielparametre;produire une liste des
code_parametreassociés à des analyses mais absents du référentiel ;produire une table des analyses “orphelines”, c’est-à-dire qui ne correspondent à aucun prélèvement.
Résultats attendus :
Résultat attendu pour les jointures entre analayse, prelevement et station
## Rows: 153,497
## Columns: 22
## $ code_analyse <int> 5186581, 280131, 1576225, 799894, 472800, 27671…
## $ code_laboratoire <dbl> NA, 292, NA, NA, 292, NA, NA, NA, NA, NA, NA, N…
## $ code_prelevement <int> 37593, 7715, 15517, 9566, 8332, 26792, 35625, 1…
## $ code_parametre <dbl> 1216, 1668, 1185, 1217, 1907, 1945, 1673, 1234,…
## $ code_fraction_analysee <int> 23, 23, 23, 23, 23, 23, 23, 23, 23, 23, 23, 23,…
## $ resultat_analyse <dbl> 0.007, 0.050, 0.040, 0.050, 0.260, 0.020, 0.010…
## $ code_remarque <int> 10, 2, 2, 2, 1, 10, 10, 10, 10, 10, 10, 10, 2, …
## $ limite_detection <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
## $ limite_quantification <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
## $ code_intervenant <fct> NA, 104, NA, NA, 104, NA, NA, 53, NA, 44, 49, 4…
## $ code_reseau <fct> OSUR, OSUR, FREDON, OSUR, OSUR, OSUR, OSUR, ARS…
## $ code_station <chr> "04153800", "04130000", "04132500", "04214000",…
## $ date_prelevement <chr> "2014-09-16", "2003-08-05", "2008-09-01", "2007…
## $ code_support <int> NA, 3, NA, NA, 3, NA, NA, 3, NA, 3, 3, 3, NA, N…
## $ libelle_station <chr> "MOZEE à CHANTONNAY", "MAYENNE à DAON", "MAYENN…
## $ date_creation <chr> "1900-01-01", "1900-01-01", "1900-01-01", "1900…
## $ source <chr> "AELB", "AELB", "AELB", "AELB", "AELB", "AELB",…
## $ code_masse_eau <chr> "GR1950", "GR0460c", "GR0460c", "GR0121", "GR04…
## $ code_entite_hydro <chr> "N3036200", "M---0090", "M---0090", "J78-0300",…
## $ code_troncon_hydro <chr> "N3036200", "M3620090", "M3910090", "J7800300",…
## $ code_commune <chr> "85051", "53089", "49214", "44036", "53017", "5…
## $ annee <dbl> 2014, 2003, 2008, 2007, 2005, 2006, 2013, 2009,…Résultat attendu pour le nombre d’analyses réalisées sur des molécules (code_parametre) présentes dans le référentiel parametre :
## [1] 194382Résultat attendu pour la liste des code_parametre associés à des analyses mais absents du référentiel :
## [1] 1261 1206 1673 1234 1686 1757 1666 1149 1865 1888 1225 1830
## [13] 1506 1136 1218 1199 1664 7097 1913 1680 5526 1235 1533 1266
## [25] 1155 1877 1215 1209 1126 1208 1189 9052 1526 1256 1903 1905
## [37] 1866 1895 1171 1130 2027 2046 1104 1233 1193 2057 1890 1173
## [49] 1148 1150 1103 2737 2047 1490 1464 1101 1254 1698 1540 1870
## [61] 2912 1287 1159 1142 1257 1213 1812 1700 1194 1432 2546 6260
## [73] 1289 1697 5537 1694 2074 1699 1951 2751 6398 1806 1228 2678
## [85] 1814 1832 2806 2015 1809 1682 1742 3159 2807 1214 1402 1670
## [97] 5921 0 1210 1276 1153 2028 1094 1968 6483 1124 1119 1891
## [109] 1127 1139 1954 1503 1523 1887 1147 1522 1743 1972 1966 1763
## [121] 2012 2976 1259 1701 1102 1231 1940 1238 2024 5416 1860 5760
## [133] 1501 1942 6824 1280 1502 2951 1112 1975 1943 1336 1711 1716
## [145] 1930 1764 1655 1283 1615 1976 1953 7057 2010 1805 2950 2930
## [157] 1546 2565 5488 1880 1774 1684 1525 1959 1616 1592 1587 2769
## [169] 2545 2750 2069 5638 2664 1702 1720 1538 1636 2984 5581 2913
## [181] 6261 6856 1091 1875 7500 1090 1649 1614 1548 7345 2872 2987
## [193] 1250 12143 2021 1274 1629 1465 2609 3209 1642 2748 1643 2744
## [205] 1969 1869 7342 1591 1251 1647 2749 1116 2986 2066 1469 1388
## [217] 2081 1245 2889 5545 1628 1648 2919 1595 1613 1957 1590 1955
## [229] 1089 2929 2536 1160 3283 1282 1387 1458 7522 1241 3160 2732
## [241] 1275 1651 1382 1106 1115 12098 1588 1468 1612 2916 1624 1594
## [253] 12099 1032 1627 2733 1243 2734 1921 1273 2915 6384 1272 1117
## [265] 1278 1653 1731 1586 1396 2887 1286 2770 5499 1305 1471 1650
## [277] 1625 1645 1593 2868 1486 1730 1161 7150 2590 2032 1626 1589
## [289] 1249 1386 1242 2537 1244 1644 1497 1622 2568 1577 1641Résultat attendu pour la table des analyses “orphelines”, c’est-à-dire ne correspondent à aucun prélèvement :
## Rows: 84,535
## Columns: 9
## $ code_analyse <int> 3854590, 1812211, 3124130, 4245401, 3684301, 38…
## $ code_laboratoire <dbl> NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA,…
## $ code_prelevement <int> 32031, 17208, 28512, 33302, 31986, 32146, 17640…
## $ code_parametre <dbl> 2025, 1261, 2045, 2978, 2018, 1190, 1206, 1911,…
## $ code_fraction_analysee <int> 23, 23, 23, 23, 23, 23, 23, 23, 23, 23, 23, 23,…
## $ resultat_analyse <dbl> 2e-02, 5e-02, 1e-02, 1e+01, 1e-02, 2e-02, 2e-02…
## $ code_remarque <int> 10, 10, 10, 10, 133, 10, 10, 10, 10, 10, 10, 10…
## $ limite_detection <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…
## $ limite_quantification <dbl> 0, 0, 0, 0, 10, 0, 0, 0, 0, 0, 0, 0, 0, 0, 10, …